1. 서 론
2. 재료 및 방법
Prorocentrum minimum 배양 및 실험 조건
광합성 지표 분석
유전자 구조 및 계통수 분석
RNA 추출 및 cDNA 합성
Polymerase Chain Reactions (PCRs) 및 클로닝
Quantitative Real-time Polymerase Chain Reactions (qRT- PCRs)
통계 분석
3. 결 과
와편모조류 Prorocentrum minimum의 CAs 유전자 특성 분석
pH에 의한 광합성 효율 변화
CA 및 광합성 연관 유전자 발현 분석
4. 고 찰
5. 결 론
1. 서 론
육상식물의 일부는 이산화탄소(CO2) 농축 메커니즘(Carbon Concentrating Mechanism, CCM)을 통해 세포 내 이산화탄소를 축적한다. 수생 광합성 생물의 CCM은 녹조류 Chlamydomonas reinhardtii에서 처음 보고되었으며(Badger et al. 1980), 다양한 생물학적·환경적 요인에 의해 효율이 조절된다(Beardall and Giordano 2002; Raven et al. 2005). CCM에 영향을 미치는 요인에는 루비스코(Ribulose-1,5-bisphosphate carboxylase-oxygenase, RuBisCO)와 CO2 친화도(Hopkinson et al. 2014), pH (Mangan et al. 2016), 용존탄소(Kim et al. 2012), 수온(Costache et al. 2013), 광조건(Gonçalves et al. 2014) 등이 있다. 최근 대기 중 CO2 농도의 증가가 해양 산성화로 이어지면서, pH 변화에 따른 해양 광합성 생물의 CO2 소비 효율이 주목받고 있다(Harvey 2008). 이산화탄소 증가로 인한 해양 산성화는 해양 생물에게 스트레스 요인으로 작용하여 생리적 변화를 초래하는데(Pörtner et al. 2004), 특히 식물플랑크톤의 광합성 및 탄소 고정 효율을 저해하는 것으로 보고되었다(Raven et al. 2014). 이는 생물 생존을 위협하므로(Anthony et al. 2008; Bobeszko 2017), 해양 산성화에 따른 해양 광합성 생물의 CCM 효율 및 CO2 대사에 대한 저해를 시사한다.
CO2 농도가 낮은 해양 환경에서 해수의 용존유기탄소(Dissolved Organic Carbon, DOC)는 CCM에 의해 CO2 로 전환되어 세포 내에 효율적으로 포집된다(Badger et al. 2002; Badger 2003). 포집된 CO2는 세포 성장을 위한 기질로 사용되며, 이때 세포 외부의 DOC를 세포 내로 수송하는 무기탄소 수송단백질(energy-dependent Ci transporter)과 탄소 무수화효소(Carbonic Anhydrase, CA)가 주로 관여한다(Fukuzawa et al. 2000; Moroney and Ynalvez 2007; Gee and Niyogi 2017). CA는 대부분의 생물체 내에서 HCO3- + H+ ⇔ CO2 + H2O 반응을 촉매하여 CO2 흡수, pH 균형 유지, 기체 교환 및 세포 내부로의 CO2 수송 등을 수행한다(Henry 1996; Lindskog 1997; Badger 2003; Boron 2004; Alvizo et al. 2014). 특히 광합성 생물의 CA는 RuBisCO 주위에 CO2를 포화시키는데(Badger and Price 1994), 이는 CO2에 대한 RuBisCO의 친화도를 높이는 동시에 CO2에 대한 O2의 경쟁적 저해를 막아 탄소 고정 효율을 향상시킨다(Yamano and Fukuzawa 2009; Griffiths et al. 2017; Wang et al. 2017).
CA는 아미노산 서열의 유사성에 따라 alpha, beta, gamma 등의 CA family로 분류되며, 세포 내 분포에 따라 세포 외 CA (extracellular CA, eCA)와 세포 내 CA (intracellular CA, iCA)로 나뉜다(Lindskog 1997). 세포 표면에 위치한 eCA는 HCO3-와 CO2 사이의 상호 전환을 비교적 느린 속도로 촉매하고, 식물플랑크톤이 수계에서 CO2를 획득하는 데에 중요한 역할을 수행한다(Kimpel et al. 1983; Coleman and Grossman 1984; Aizawa and Miyachi 1986; Mustaffa et al. 2017). 반면 iCA는 세포질, 미토콘드리아, 엽록체에 존재하며, 세포 내 위치에 따라 전환시키는 탄소 분자의 종류가 다르다(Aizawa and Miyachi 1986; Merrett et al. 1996; Moroney and Somanchi 1999). CA는 고등 식물과 진핵세포성 조류에서 연구되었는데, 특히 미세조류는 다른 광영양생물보다 탄소 고정에 있어 CA 의존도가 높다고 보고되었다(Tang et al. 2011; Boone et al. 2013; DiMario et al. 2018).
수생 광합성 생물은 종에 따라 각기 다른 형태의 RuBisCO (Form I, II)를 갖는다. 이들 모두 산소성 광합성에 사용되지만, Form I의 탄소 고정 효율이 Form II보다 높다(Stein and Felbeck 1993; Raven and Beardall 2003; Beardall and Raven 2016; Rickaby and Hubbard 2019). 대부분의 해양 광합성 생물은 Form I의 RuBisCO를 갖는 반면, 와편모조류의 경우 일반적으로 Form II를 갖는다(Rickaby and Hubbard 2019). 해양 와편모조류가 효율이 낮은 RuBisCO (Form II)를 사용함에도 불구하고, 전 세계의 다양한 해양 환경에 적응하고 성장 조건이 충족되면 대량 증식하여 적조를 유발한다(Bercovici and Vellekoop 2017). 이는 와편모조류가 세포 내 특이적인 CO2 수송 및 전환 메커니즘을 통해 낮은 효율의 RuBisCO 활성을 보완한다는 것을 제시한다(Leggat et al. 2002). 하지만 현재까지 광합성 조류의 CCM에 대한 연구는 녹조류 C. reinhardtii에 집중된 반면, 와편모조류를 대상으로 한 연구는 부족하다. 와편모조류는 해양 미세조류 먹이그물에서 다양한 위치를 차지하고 있으며, 특히 Symbiodinium의 경우 산호와 공생하여 산호의 환경 스트레스 내성과 회복에 직접적인 영향을 미친다(Jeong et al. 2010; Suggett et al. 2017). 산호의 탄소 대사에 관여하는 Symbiodinium sp.의 CA 연구가 선행되었으나(Bobeszko 2017), 유영 와편모조류를 대상으로 한 CA 유전자의 발굴 및 전사 조절에 대한 연구는 거의 수행되지 않았기 때문에, 다양한 와편모조류에 대한 CA 유전자의 분자적 및 생화학적 연구가 필요하다.
본 연구는 해양 와편모조류 Prorocentrum minimum에서 3 개의 CA 유전자를 발굴하고 유전적 특성과 분자계통학적 특성을 분석하였다. 또한, pH 8의 해수에 적응된 P. minimum을 다양한 pH 변화(pH 6–10)에 6시간 동안 노출시킨 후, 세포의 광합성에 미치는 영향, 광합성 및 CA 유전자의 상대적 발현을 파악하였다. 실험 종 P. minimum은 전세계 해안 및 기수 지역에 분포하며, 다양한 환경 스트레스 실험에 사용된 해양 모델 생물이다(Wang et al. 2018; Kim et al. 2021). 이를 통해, 해양의 pH 변화가 해양 와편모조류에 미치는 영향을 파악하였다.
2. 재료 및 방법
Prorocentrum minimum 배양 및 실험 조건
Prorocentrum minimum 배양주(D-127)는 한국해양미세조류은행(부경대학교, 부산)에서 분양받았다. 배지는 GF/C filter paper (Cat. No. 1822-047)로 여과한 해수에 bicine (Cat. No. B8660; Sigma-Aldrich)을 20 mM이 되도록 첨가하고, 교반기에서 충분히 녹인 뒤 5.0 M NaOH와 1.0 M HCl를 사용하여 pH 8로 조절하였다(Rost et al. 2006). 121°C에 15분간 멸균 후 냉각하여 silica를 제외한 f/2 배지(Guillard and Ryther 1962)를 제조하였다. f/2 배지에 P. minimum을 접종 후, 7일간 매일 배지의 pH를 8로 조절하였다. 배양주는 20 °C, 12:12시간 명:암 주기, 65 μmol photons/m2/s의 빛 조건에서 배양하였다. 지수기의 P. minimum (5 × 104 cells/mL) 배양주 200 mL를 300 mL Duran flask에 소분하고, 5.0 M NaOH와 1.0 M HCl을 이용하여 각 샘플의 pH를 pH 6, 7, 8, 9, 10으로 조정하였다. 수중에 우세한 용존탄소 화학종들의 분포는 pH에 의해 영향을 받는데(화학 평형의 법칙), H2CO3 분포가 높은 pH 6 그리고 CO32- 분포가 높은 pH 10까지 관찰하였다(Stumm and Morgan 2012). pH 측정은 SevenCompactTM pH meter S220 (Mettler Toledo Korea, Seoul, Korea)을 사용하였다.
광합성 지표 분석
배지의 pH 변화가 P. minimum의 광합성에 미치는 영향을 파악하기 위해, pH 조정 0, 1, 2, 3, 6시간 후 광합성 지표를 측정하였다. 각 샘플을 2 mL씩 표본병에 옮겨 담고, 20°C에서 20분간 암반응을 제공하였다. Handy Plant Efficiency Analyser fluorometer (Hansatech Instruments Ltd., Norfolk, UK)를 사용하여 광합성 지표(Fv/Fm, PItotal, ABS/RC, TRo/RC, ETo/RC, DIo/RC)를 각 시간대별로 분석하였고, 모든 샘플은 3회 반복하여 측정하였다.
유전자 구조 및 계통수 분석
P. minimum의 CA 유전자는 차세대염기서열분석(Next- generation sequencing, NGS)을 이용한 Expressed Sequence Tags (EST) 데이터에서 선별하였다. PmCA 단백질의 보존 서열은 PROSITE (http://prosite.expasy.org/), National Center for Biotechnology Information (NCBI)의 Conserved Domains (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb. cgi)에서 분석하였고, signal peptide (SP) 및 단백질 위치 예측은 Phobius (http://phobius.sbc.su.se/), TargetP-2.0 (https:// services.healthtech.dtu.dk/service.php?TargetP-2.0), WoLF PSORT (https://wolfpsort.hgc.jp/)에서 분석하였다(Horton et al. 2007). 또한 단백질 3차 구조 모델링은 SWISS-MODEL (http://www.expasy.org/swissmod/SWISS-MODEL.html)에서 수행하였다. NCBI의 Basic Local Alignment Search Tool (BlastP)에서 PmCA1-3과 상동성이 높은 아미노산 서열을 선별하였으며, MEGA X의 maximum likelihood (ML) 방법을 기반으로 계통수를 작성하였다(Kumar et al. 2018).
RNA 추출 및 cDNA 합성
각 샘플은 3,000 × g에서 10분간 원심분리하여 수확하였고, 즉시 액체 질소에서 동결시킨 후 -80℃에서 보관하였다. 보관된 샘플은 동결-융해 과정으로 물리적으로 파쇄하였고, Mini-Bead beater (BioSpec Products Inc, Bartlesville, OK, USA)에 zirconium beads (지름 0.1 mm)를 사용하여 세포를 균질화 하였다. 이후 제조사의 프로토콜을 기반으로 TRizoL (Invitrogen, Carlsbad, CA, USA) 시약을 사용하여 mRNA를 추출하였고, 추출한 mRNA를 Mini Spin Columns of RNeasy Mini Kit (Qiagen, Valencia, CA, USA)로 정제하였다. mRNA의 농도는 Agilent 2100 Bioanalyzer (Aglient, Santa Clara, CA, USA)로 확인하였다.
Complementary DNA (cDNA)는 TOPscript™ cDNA Synthesis Kit (Enzynomics, Daejeon, Korea), random hexamer 그리고 oligo (dT)18를 사용하여 합성하였다. 합성된 cDNA는 nuclease-free 증류수로 3배 희석하여, 상대적 유전자 발현 분석의 샘플로 활용하였다.
Polymerase Chain Reactions (PCRs) 및 클로닝
PmCAs의 cDNA 서열은 각 유전자에 특이적인 프라이머(Table 1)를 사용하여 polymerase chain reactions (PCRs) 증폭 후 클로닝하였다. PCRs은 TaKaRa Ex Taq® (Cat. No. RR001A; Takara, Seoul, Korea)을 사용하였다. PCR 혼합물에는 10× Ex Taq Buffer 2 µL, dNTP Mixture 2 µL, 희석된 cDNA 샘플 1 µL, 프라이머 각 1 µL, TaKaRa Ex Taq 0.2 µL 그리고 증류수 12.8 µL를 첨가하였다. PCRs은 아래와 같은 조건으로 진행하였다: 95°C, 3분; (95°C, 30초; 55°C, 30초; 72°C, 1분) × 30회; 72°C, 5분. 증폭된 PCRs 생성물은 전기영동으로 적합한 사이즈의 밴드를 잘라 DNA Gel Extraction S & V Kit (Bionics, Seoul, Korea)로 정제하였다. 정제된 PCRs 생성물은 TOPcloner™ TA kit (Enzynomics)로 클로닝하고, 이를 시퀀싱하여 cDNA 서열을 확보하였다. 확보된 서열은 Sequencher 5.1 (Gene Codes, Ann Arbor, MI, USA)로 조합하여 EST 데이터에서 얻은 서열과 비교였다.
Table 1.
Primer information used for quantitative real-time polymerase chain reactions (qRT-PCRs) and cloning
| Gene | Primer | Sequence 5´→3´ | Reference |
| PmCA1 | PmCA6001-B-F1 | AAGAGGCTTGACGTGGATTTCA | qRT-PCRs |
| PmCA6001-B-R1 | CCTCTATCTGCGTTTTCCTCGT | ||
| PmCA1-F0 | CGAAGTTCGGCAGAGAGATGG | Cloning | |
| PmCA1-R0 | GTCTCCGCAGCATCGCATCG | ||
| PmCA1-F1 | GAGTCAGGGCGTCTACCACCA | ||
| PmCA1-R1 | TGATAGCTTGCCCACATGCCTC | ||
| PmCA2 | PmCA11945-A1-F2 | GAAGTTGGCGTTCGATATCTGC | qRT-PCRs |
| PmCA11945-A1-R2 | GACGGGATGATAGCTGACTCTG | ||
| PmCA2-F0 | ATCGACATGCCTCTGACCTCTGC | Cloning | |
| PmCA2-R0 | CTCGGACCCTGAGATCACTCGG | ||
| PmCA2-F2 | ACGTTACCGACCATGCGGAC | ||
| PmCA2-R2 | GCACGTCGCGGTTGAAGGTA | ||
| PmCA3 | PmCA14014-A2-F3 | GCCCAAGTACGACTTCTACCTC | qRT-PCRs |
| PmCA14014-A2-R3 | GAACGTCGCGAACATCACAAAG | ||
| PmCA3-F0 | GCGAGAGGCAGCAAGATGTGG | Cloning | |
| PmCA3-R0 | GCCACGGCTCAGAGAATGTTCG | ||
| PmCA3-F2 | GAAGTTCGAGCTGCCCGTGAAC | ||
| PmCA3-R2 | CCAAGGAAAACAGCGCGGAGA | ||
| PmatpB | PmatpB-F | CAGCAGACGACCTAACGGATC | Guo et al. 2017 |
| PmatpB-R | CCTTTGCTTGCCAGGTTCCTG | ||
| PmrbcL | PmrbcL-F | AGTTGTGGAAGGCAGGCACCTACG | Guo et al. 2017 |
| PmrbcL-R | GCGTCTTCGCGTACTCGACGAC | ||
| PmTUA | PmTUA-F | GCGTGCTGCATGATGTATCGTG | Guo et al. 2014 |
| PmTUA-R | ATCCGGTAGGGCACCAATCAAC |
Quantitative Real-time Polymerase Chain Reactions (qRT- PCRs)
qRT-PCRs은 TOPreal™ qPCR 2 × PreMIX SYBR Green Kit (Enzynomics)와 CFX96 Real-Time PCR Detection System (Bio-Rad, Hercules, CA)를 사용하였다. PCR 혼합물에는 2× PreMIX SYBR Green 10 µL, 희석된 cDNA 샘플 1 µL, 프라이머 각 0.5 µL, 그리고 증류수 7 µL를 첨가하였다. qRT-PCRs은 아래와 같은 조건으로 진행하였다: 50°C, 4분; 95°C, 10분; (95°C, 10초; 60°C, 15초; 72°C, 15초) × 40회; 72°C, 15초. 증폭의 특이성은 샘플을 65°C에서 95°C로 가열하여 생성된 용융 곡선(melt curve)으로 결정하였다. 각 유전자에 특이적으로 반응하는 프라이머를 제작하고, 표준 곡선을 작성하여 90–110% 사이의 효율을 보이는 프라이머를 선별하였다(Table 1). 상시 발현 유전자 Alpha-tubulin (PmTUA)을 internal reference gene으로 설정하였고, PmCA1, PmCA2, PmCA3, PmatpB, PmrbcL의 유전자 발현율(∆Cq)을 계산하였다. 대조군(pH 8)의 평균 ∆Cq 값을 토대로 각 처리군의 상대적 유전자 발현율(∆∆Cq)을 계산하였다. 모든 샘플은 3회 반복으로 실험하였다.
통계 분석
평균 및 표준 오차는 3회 반복된 각 샘플 결과를 대상으로 계산하였다. pH 변화가 P. minimum에 미치는 영향을 조사하기 위해 SPSS20 통계 패키지(SPSS Inc., Chicago, IL, USA)를 사용하였고, 분산분석(Analysis of Variance, ANOVA)을 진행한 후 Duncan's multiple range test (p < 0.05)를 수행하여 대조군과 실험군의 유의성을 분석하였다. 유의성은 *p < 0.05, **p < 0.01, ***p < 0.001로 표시하였다.
3. 결 과
와편모조류 Prorocentrum minimum의 CAs 유전자 특성 분석
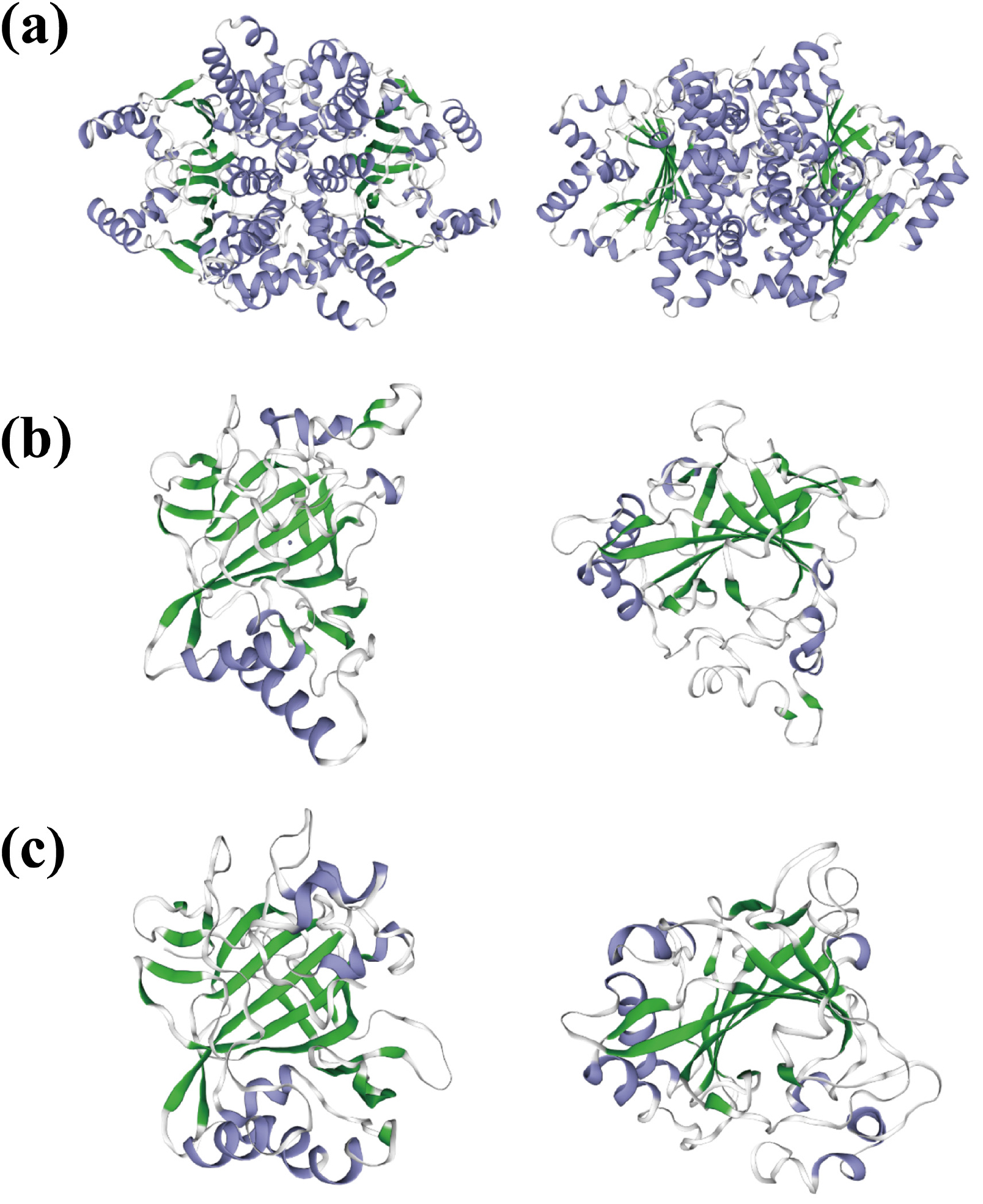
해양 와편모조류 P. minimum (D-127)의 EST 데이터로부터 3 개의 PmCAs 유전자(PmCA1; comp6001_c0_seq1, PmCA2; comp11945_c0_seq1, PmCA3; comp14014_c0_ seq1)를 선택하였다. PmCA1-3 cDNA유전자 서열의 길이는 각각 830 bp, 2,114 bp, 2,792 bp이며, open reading frame (ORF) 길이는 각각 231 aa, 558 aa, 879 aa이다. 보존된 도메인 분석 결과, PmCA1 (1-193 aa)에는 beta-CA 도메인이, PmCA2 (248-473 aa)와 PmCA3 (576-799 aa)에는 alpha-CA 도메인이 확인되었다. 또한, PmCA1에는 4 개(Cys36, Asp38, His92, Cys94), PmCA2에는 3 개(His319, His321, His338), PmCA3에는 3 개(Thr650, His652, Gln669)의 zinc (Zn) binding site가 존재한다. SP 분석 결과, PmCA1은 세포질 내 수용성(soluble) 단백질, PmCA2는 엽록체 막 관통 단백질, PmCA3는 세포 외 막 관통 단백질로 예측된다.
PmCAs의 단백질 3차 구조는 SWISS-MODEL에서 Global Model Quality Estimate (GMQE)와 아미노산 서열 일치율(Identity)이 가장 높은 모델을 기반으로 예측하였다(Fig. 1). PmCA1은 주형 모델 4o1k.1.A와 53.2% 일치하였고, 사량체(homo-tetramer) 중합 모델로 확인되었다. PmCA2-3은 각각 주형 모델 1y7w.1.A, 4pxx.1.A와 23.3%, 37.35% 일치하였고, 단량체(monomer) 모델로 확인되었다.
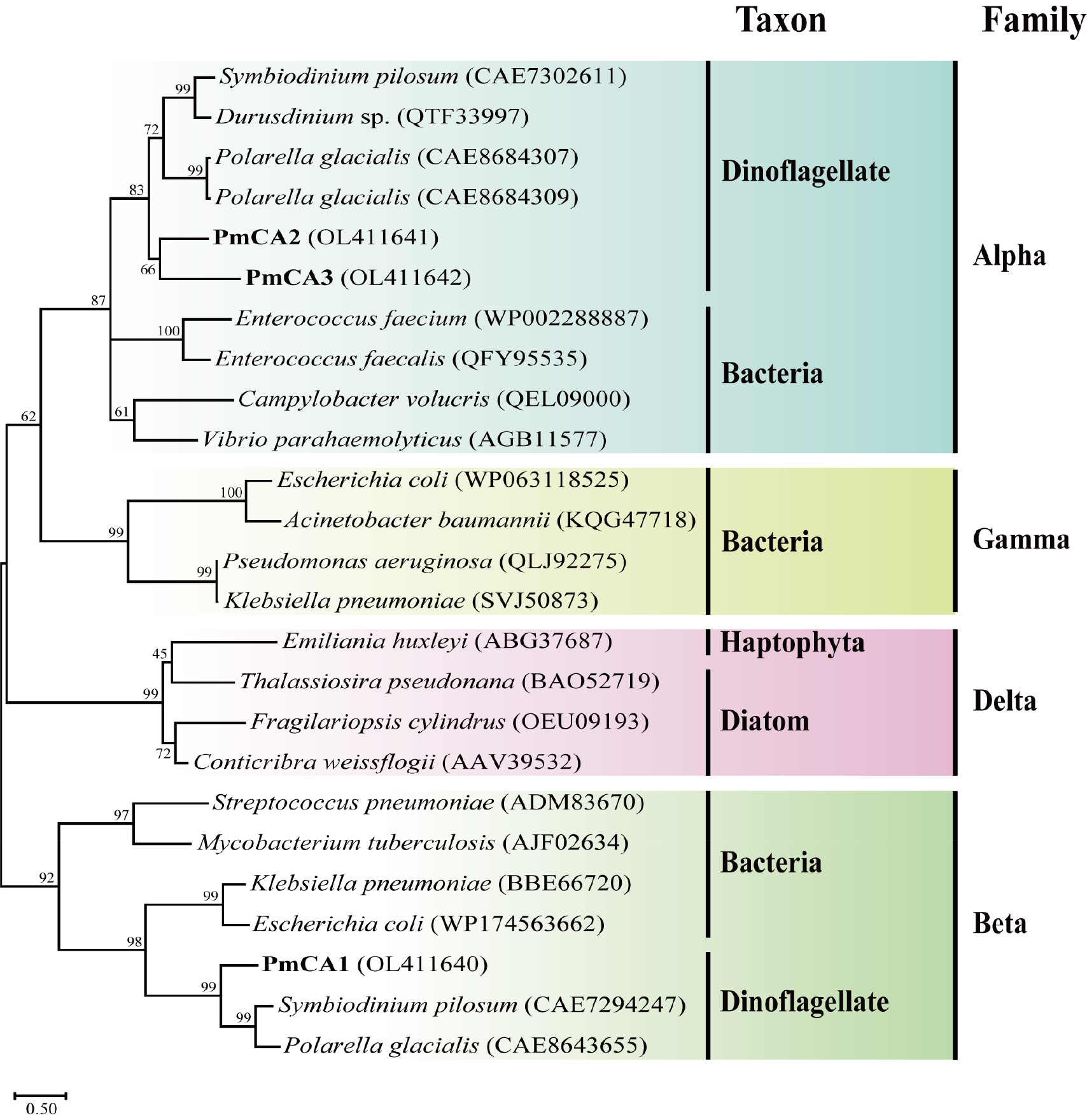
ML 계통수 기반으로 PmCA1-3과 다른 조류 및 박테리아 CAs와의 근연 관계를 확인하였다(Fig. 2). 보존 도메인 결과와 동일하게 PmCA1은 beta-CA superfamily, PmCA2 -3은 alpha-CA superfamily에 속하였다. 이들은 기존에 알려진 와편모조류 CA와 가장 높은 유사도를 보였으며, PmCA1은 Polarella glacialis (GenBank no. CAE8643655)와 59.2%, Symbiodinium pilosum (CAE7294247)과 56.9%의 상동성을, PmCA2는 Symbiodinium necroappetens (CAE 7659234)와 45.6%, Symbiodinium natans (CAE7023010)와 44.6%의 상동성을 보였다. 마지막으로 PmCA3는 P. glacialis (CAE8688696)와 39.1%, Symbiodinium microadriaticum (CAE7389539)와 38.9%의 상동성을 보였다.
pH에 의한 광합성 효율 변화
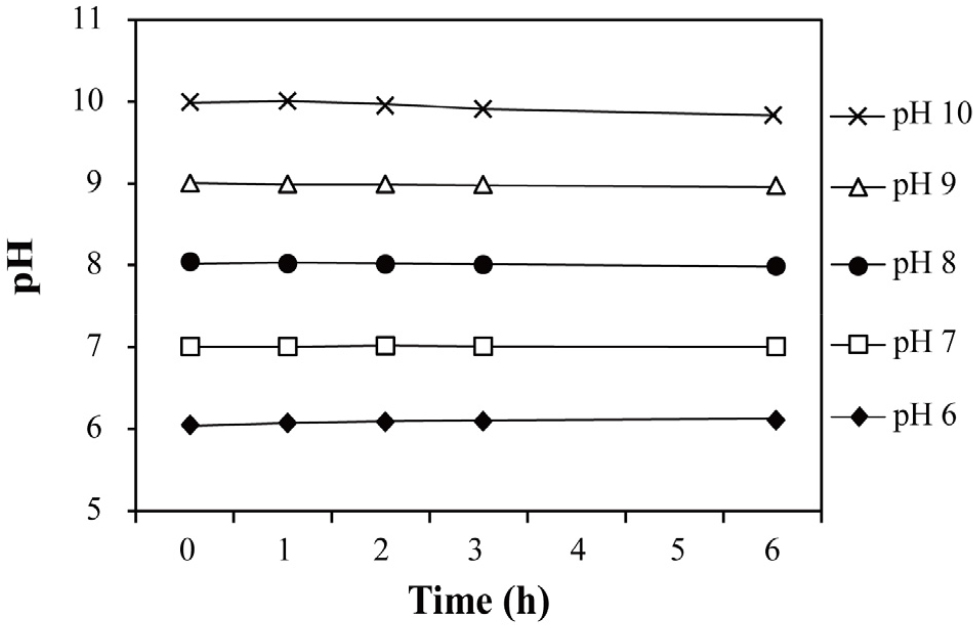
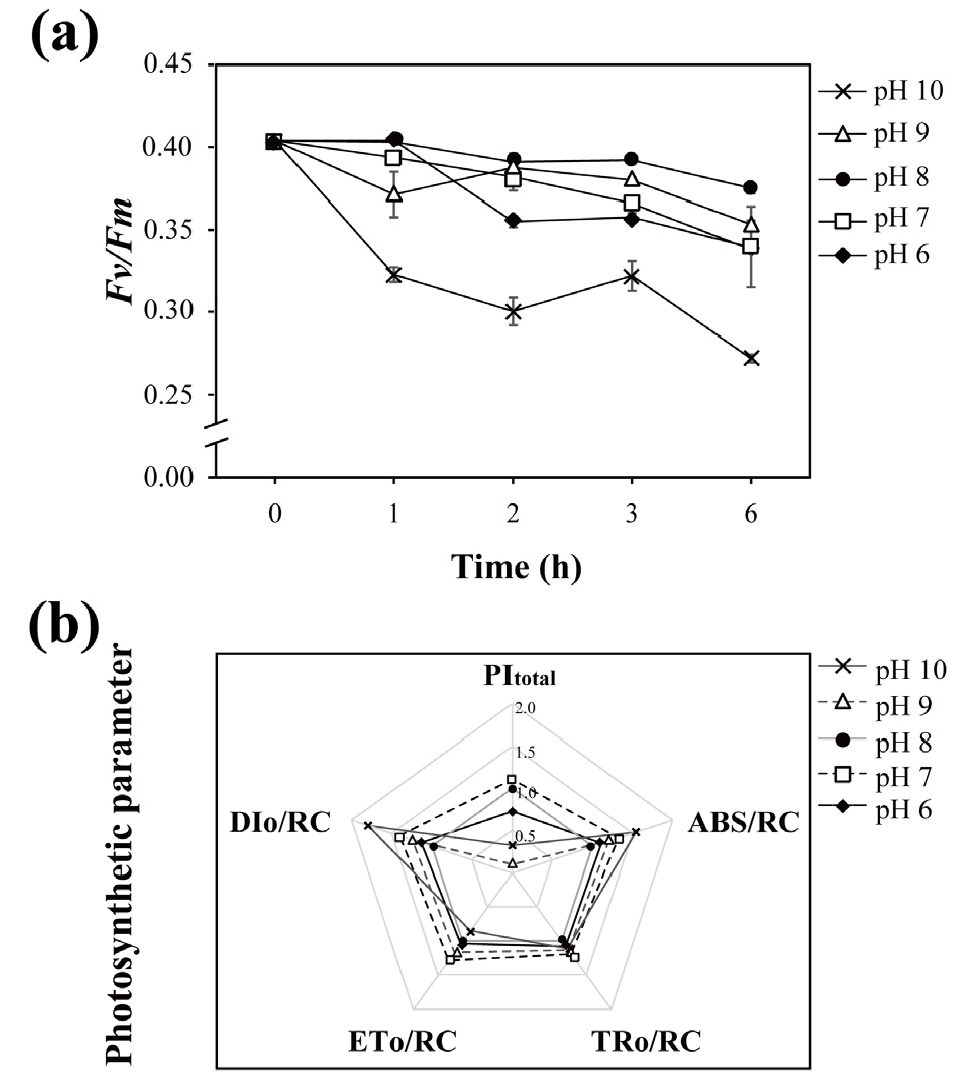
pH 변화를 관찰하기 위해 5 개로 소분한 배양주의 pH를 각각 6, 7, 8, 9, 10으로 조정한 후 0, 1, 2, 3, 6 시간마다 pH를 측정하였다(Fig. 3). 또한, pH 변화에 따른 광합성 효율(Fv/Fm)과 광합성 지표(PItotal, ABS/RC, TRo/RC, ETo/ RC, DIo/RC)를 측정하였다(Fig. 4). 대조군 pH 8과 비교했을 때, Fv/Fm은 pH 변화 후 전반적으로 감소하는 경향을 보였으며, 그 중 pH 10으로 변경 1시간 후부터 값이 유의하게 감소하였다. 2시간 및 6시간 이후 대조군 대비 각각 26%, 29% 감소하였다. 광합성 지표는 각 조건에서 얻은 값을 대조군(pH 8)으로 나누어 상대적 변화율을 계산하였다. 그 결과, PItotal은 pH 7을 제외한 모든 pH 조건에서 대조군에 비해 감소하였고, pH 9에서 최대 68% 감소하였다. ABS/RC, TRo/RC, ETo/RC, DIo/RC는 대부분의 pH 조건에서 대조군보다 증가하였고, 특히 ABS/RC와 DIo/ RC는 pH 10에서 각각 1.5배, 1.8배 증가하였다. 반면 pH 10 조건에서의 ETo/RC는 대조군 대비 15% 감소하였다.
CA 및 광합성 연관 유전자 발현 분석
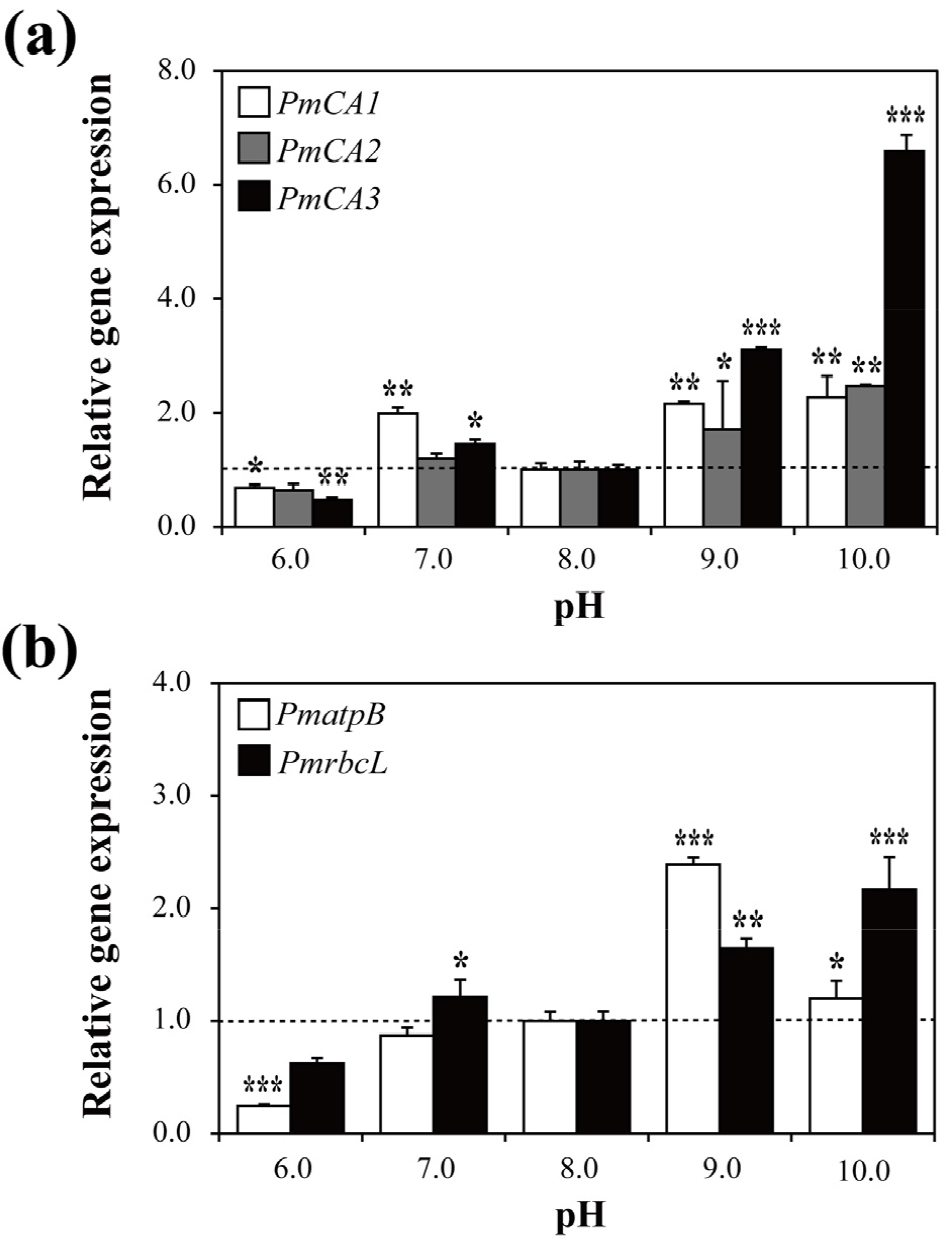
pH 변화 6시간 이후, 3 개의 PmCAs 유전자와 2 개의 광합성 연관 유전자(PmatpB, PmrbcL)의 상대적 유전자 발현율을 분석하였다(Fig. 5). 전반적으로 모든 유전자의 상대적 발현율은 pH 6에서 감소하였고, pH가 증가할수록 높아지는 양상을 보였다. pH 6에서 PmCA1 (p < 0.05)과 PmCA3 (p < 0.01)의 유전자 발현율이 통계적으로 유의하게 감소하였다. 반면, pH 10에서 PmCA1과 PmCA2의 상대적 유전자 발현율은 각각 2.2배(p < 0.01)와 2.3배(p < 0.01) 증가하였다. 특히, PmCA3의 pH 9, pH 10 조건에서 유전자 발현율은 각각 3.1배, 6.6배(p < 0.001) 증가하였다.
PmatpB의 상대적 유전자 발현율은 pH 6에서 유의하게 감소하였고(0.2배, p < 0.001), pH 9 (2.3배, p < 0.001)와 pH 10 (1.2배, p < 0.05)에서 유의하게 증가하였다. PmrbcL은 pH 6에서 유의한 변화가 없었다. 반면 pH 9 (1.7배, p < 0.01) 와 pH 10 (2.2배, p < 0.001)에서 유의하게 증가하였다.
4. 고 찰
Prorocentrum minimum은 간독소(hepatototoxin) 및 설사성 패독(diarrhetic shellfish toxins)을 합성하는 대표적인 종으로(Shimizu 1978), 대개 연안에서 적조를 발생시켜 해양 생물 및 사람에게 악영향을 미친다(Heil et al. 2005; Anderson et al. 2012). P. minimum은 전세계 해안에 분포하며(Heil et al. 2005), 다양한 염분, 빛, 온도, 낮은 영양 조건에 적응 및 생존한다(Tyler and Seliger 1981; Harding 1988; Harding and Coats 1988; Grzebyk and Berland 1996). 이와 같이 P. minimum은 다양한 환경 변화에 생리적으로 유연하게 대응할 수 있어(Heil et al. 2005), 와편모조류 중 대표적인 실험 모델 생물로 사용되고 있다. 지금까지 이 종을 대상으로 한 스트레스 노출(중금속, 제초제, 염소)에 따른 생리적 및 분자적 지표(molecular biomarker) 연구가 많이 진행되었다(Ebenezer and Ki 2013a, 2013b). 따라서 P. minimum을 대상으로 pH 변화가 해양 와편모조류의 CCM에 미치는 영향을 조사하는 것이 적합하다고 사료된다. 본 연구는 P. minimum을 통해 비교적 선행 연구가 미흡한 와편모조류의 CA 구조 및 생화학적 특성 파악하고, pH에 따른 CA의 유전자 발현 변화를 파악하였다.
alpha- 및 beta-CA는 active site와 Zn2+의 반응으로 활성화된다(Xu et al. 2008). alpha-CA는 3 개의 histidine 잔기와 OH-, Zn2+이 반응한다. beta-CA는 open active site를 갖는 type I과 closed active site를 갖는 type II로 나뉜다. beta-CA는 2 개의 cysteine, 1 개의 histidine이 Zn2+과 결합하고, 네 번째 Zn ligand에 OH- (type I) 또는 aspartate 잔기가(type II) 결합한다(Supuran 2016; Kupriyanova et al. 2017). PmCA1은 beta-CA의 Zn binding site (Cys36, Asp38, His92), 그리고 active site (Gln28, Asp38, Ser39, Arg40)를 가졌으며(Rowlett et al. 2010), Emameh et al. (2014)이 제시한 beta-CA의 보존된 서열(Cys-Xaa-Asp- Xaa-Arg)이 확인되었다. 또한, beta-CA의 경우 단량체인 alpha-CA와 달리 이량체, 사량체 혹은 팔량체를 형성하여 기능을 수행하는데, PmCA1은 사량체로 확인되었다(Capasso and Supuran 2015). PmCA2는 3 개의 Zn binding site (His319, His321, His338)를 가졌으며, Zn2+과 물 분자의 친핵성도(nucleophilicity)를 높이는 Thr424와 Glu325가 확인되었다(Smith and Ferry 2000; Capasso and Supuran 2015). PmCA3은 2 개의 active site와 Zn binding site (His128, Val130, Phe14; Thr650, His652, Gln669)가 있고, 마찬가지로 Thr749와 Glu656가 확인되었다.
SP는 3 개의 고유 도메인으로 나뉘며, 양전하로 하전된 아미노 말단기(n-region), 중심부의 소수성 부위(h-region), 그리고 극성 카복실 말단 도메인(c-region)가 있다(von Heijne 1990). Signal peptidase는 SP c-region의 Ala-Xaa- Ala에 작용 및 절단하여 단백질이 세포막에서 분리되도록 유도한다(van Roosmalen et al. 2004; Tuteja 2005; Driessen and Nouwen 2008). Phobius 분석 결과, PmCA2와 PmCA3에는 막 관통 부위(transmembrane region; ILLPVFGSXXL XVHVMXXYF) SP 서열이 확인되었고, 특히 PmCA3는 신호 펩타이드 가수분해 효소(signal peptidase)가 반응하는 Ala-Arg-Ala 서열을 갖는다.
P. minimum의 성장 가능 pH 범주는 pH 7.0–9.6이며, 주변 pH 변화에 적응하는 데에 약 2–3시간이 필요한 것으로 보고되었다(Hansen 2002; Berge et al. 2010). 반면, 연구종 P. minimum D-127은 산성 조건(pH 6)으로 변화 후 1시간 이내에 Fv/Fm이 감소하였다. 이는 광합성에서 양자 수득율이 산성 조건에서 1시간 이내에 감소하며, 결과적으로 광계 II에 영향을 미친다는 것을 보여준다(Strasser et al. 2000). 뿐만 아니라, 산성 조건(pH 6–7) 노출 이후 감소한 PItotal 값이 이를 뒷받침한다. PItotal은 양자 에너지 전환 효율을 근거로 한 광합성 활성 지표로, 알칼리성 환경(pH 10) 또한 P. minimum의 광합성을 저해함을 알 수 있다(Oukarroum et al. 2007).
담수 및 해수에 용존하는 CO2의 조성 및 농도는 pH에 의존적으로 변화하며, 이는 PmCAs의 상대적 발현에 영향을 미친다(Prabhu et al. 2009). 선행 연구에 따르면, 알칼리성 환경에서 탄소 고정 효율 증가를 위해 iCA의 활성이 증가하는데(Rost et al. 2006), 마찬가지로 pH 9–10 조건에 노출된 P. minimum의 PmCA1 및 PmCA2의 상대적 유전자 발현율이 유의하게 증가하였다. 게다가, Nimer et al. (1998)의 보고에 따르면, eCA의 활성은 pH가 높을수록 증가했는데, eCA에 해당하는 PmCA3의 상대적 유전자 발현율은 pH 10에서 가장 높았다. 이는 P. minimum이 HCO3-가 우세한 알칼리성 환경에 노출되었을 때, CO2 수송 및 고정을 증가시키기 위해 다양한 CA 단백질의 합성을 유도한다는 것을 암시한다(Aizawa and Miyachi 1986; Hopkinson et al. 2013).
PmatpB와 PmrbcL은 각각 틸라코이드막의 adenylpyrophosphatase (ATPase)와 RuBisCO의 소단위체를 암호화하는 유전자이다(Gillham and Boynton 1986; Klein et al. 1994; Chiang et al. 1998). ATPase는 빛 에너지로부터 ATP를 합성하는데, 이는 CO2가 탄소 화합물로 전환될 때 사용되며(Mitchell et al. 1966), RuBisCO는 CO2 고정을 돕는다(Parry et al. 2013). 두 유전자의 발현 양상은 광합성 지표와 유사한 경향을 보이며, 산성 조건(pH 6–7)에서의 유전자 발현 감소 또는 유의하지 않은 변화는 광합성 저해에 의한 영향으로 사료된다. 또한, 알칼리성 조건에서의 유전자 발현 증가는 CA의 발현 증가와 연결되는데, CA 단백질 활성 증가는 세포 내 CO2 포화를 유도하여 RuBisCO의 합성이 유도된 것으로 해석된다(Kimpel et al. 1983; Aizawa and Miyachi 1986; Badger and Price 1994).
지난 세기 동안 산업화 및 경제적 성장에 따라 화석연료의 사용이 크게 증가하였고, 대기 중 CO2 농도는 산업혁명 이전 280 ppm 대비 약 50% 증가한 410 ppm을 기록하고 있다(Widdicombe et al. 2013; IPCC 2014). 대기 중 CO2는 해양에 용해되는 즉시 빠르게 탄산염(CO₃²⁻) 및 중탄산염(HCO₃⁻)으로 전환되는데, 이는 해수의 수소 이온(H+)의 농도를 증가시킨다(Gattuso and Buddemeier 2000; Barker and Ridgwell 2012). 해수의 H+ 증가는 해양 산성화를 초래하며, 식물플랑크톤과 같은 광영양생물의 광합성 및 탄소 고정 효율을 저해한다(Raven et al. 2014). 이러한 환경에서 CCM 및 CA의 활성은 탄소 포집 효율을 향상시키고 생존에 필요한 광합성 효율을 높이기 때문에 연구의 중요성이 부각된다(So et al. 2002). 본 연구에서는 전 세계적으로 적조를 일으키는 P. minimum에서 3 개의 CA 유전자를 발굴하였고, 이들의 구조적 특징을 파악하였다. 또한, 광합성 효율, CA 유전자 및 광합성 관련 유전자의 상대적 발현을 분석하여 pH 변화가 미치는 분자적 영향을 파악하였다.
5. 결 론
탄산무수화효소(CA)는 탄소농축기작(CCM)의 핵심적인 조절인자로, 주변 pH와 이산화탄소의 조성에 영향을 받는 것으로 알려져 왔다. 본 연구에서 와편모조류 P. minimum의 3 개 신규 CAs 유전자(PmCA1-3)의 특징을 분석하고, pH 변화에 따른 PmCAs와 광합성 유전자 PmatpB, PmrbcL의 상대적 유전자 발현을 규명하였다. SP 및 계통수 분석을 통해 PmCA1은 beta family로, PmCA2와 PmCA3은 alpha family로 분류하였고, 분류에 맞게 Zn binding site를 이루는 아미노산 잔기를 확인하였다. PmCA1와 PmCA2는 세포 내 CA이며, 각각 세포질과 엽록체 막에 위치하는 것으로 파악되었다. 반면, PmCA3는 세포 외 CA로 세포막 단백질로 추정되었다. pH 변화에 따른 P. minimum의 광합성 효율은 pH 7–9에서 유사한 값을 보였으며, pH 6과 pH 10에서 유의하게 감소하였다. 대조군(pH 8)과 비교하여 모든 PmCAs 유전자 발현율은 낮은 pH에서 감소하고, pH가 높아질수록 전반적으로 증가하였다. 광합성과 관련된 PmatpB와 PmrbcL 유전자들의 발현도 PmCAs 발현과 유사한 패턴을 보였다. 본 연구결과는 해수의 pH 변화가 해양 와편모조류의 광합성 및 이산화탄소 대사에 영향을 준다는 것을 제시한다.