1. 서 론
해양생태계에서 동물플랑크톤은 가장 풍부하고 넓게 분포하는 후생동물 중 하나로, 해양 먹이망과 생화학적 순환에서 중요한 역할을 한다(Mauchline 1998; Schminke 2007). 동물플랑크톤은 기후 변화나 오염 등의 환경 변화에 빠르게 반응하여 해역의 환경지시자가 되기 때문에(Gannon and Stemberger 1978; Beaugrand et al. 2010), 동물플랑크톤 종 다양성 및 생태 연구는 환경 보호나 자원 관리에 기여할 수 있다.
동물플랑크톤에는 많은 유사종과 자매종(cryptic species)이 있고, 유생기에 형태적 특성이 뚜렷하지 않기 때문에, 종 동정을 위한 형태 분석은 시간이 많이 걸리며 전문 지식을 필요로 한다. 비교적 최근에 고안된 DNA 메타바코딩(metabarcoding)은 다양한 환경과 넓은 해역에서 채집한 동물플랑크톤을 별도의 동정 작업 없이 빠르게 분석 가능하므로(Shokralla et al. 2012; Bucklin et al. 2016), 크기가 작고 분류군이 다양한 해양 동물플랑크톤 종 동정에 유용한 분석법이다.
DNA 메타바코딩을 해양 동물플랑크톤에 활용하기 위해서는 기존의 형태 분석과의 비교 검토가 필요하다. 해양 동물플랑크톤을 대상으로 한 형태 분석과 메타바코딩의 비교 연구는 다수 보고되었으나(Lindeque et al. 2013; Abad et al. 2016; Harvey et al. 2017; Hirai et al. 2017a, 2017b; Djurhuus et al. 2018; Bucklin et al. 2019; Schroeder et al. 2020), 우리나라에서는 소수의 연구(Kim et al. 2019, 2020)만이 보고되었다. 해역에 따라 동물플랑크톤 군집의 구성과 특성이 다르므로, 우리나라 근해에서의 비교 검토가 축적될 필요가 있다.
본 연구에서는 황해에서 다수 출현하는 갑각류 중형동물플랑크톤을 대상으로 형태 분석과 메타바코딩 결과를 비교하고, 두 방법의 차이에 대해 고찰하였다. 이러한 결과는 향후 우리나라 해역의 동물플랑크톤에 대한 효과적인 생태학적 연구 방안을 찾는데 활용될 수 있을 것이다.
2. 재료 및 방법
시료 채집
2019년 4월 20일부터 24일까지 황해에 위치한 총 17개 정점(북위 35도 라인 8개, 36도 라인 4개, 37도 라인 5개)에서 중형동물플랑크톤을 채집하였다(Fig. 1). 봉고네트(망구 60 cm, 망목 200 μm)를 저층부터 표층까지 1회 복경사 예인(oblique tow)하여 형태 분석을 위해 시료를 5% 중성 포르말린으로 고정하였고, 다시 1회 반복 채집하여 DNA 메타바코딩을 위해 시료를 95% 에탄올로 고정하였다. 반복 채집 과정에서 시차가 발생하였으나, 여러 정점의 결과를 통합 하였으므로 정점 내 시료의 차이를 무시할 수 있다는 가정 하에 비교 연구를 수행하였다.
형태 분석
실험실로 운반한 시료를 분할하여 최종 300–500 개체를 해부현미경(Stemi 2000-C, Zeiss, Germany)과 광학현미경(Axioskop, Zeiss, Germany)으로 종 동정하였다. 동물플랑크톤의 종별 형태적 정보는 Chihara and Murano (1997)의 도감을 참고하였다.
DNA 메타바코딩
동물플랑크톤 시료의 전 처리를 위해 인산완충생리식염수 600 μl를 첨가하여 교반 후 6000 rpm에서 10분 동안 원심분리를 진행하였다. 원심분리가 끝난 동물플랑크톤 시료는 상층액을 제거 후 TIANamp Marine animals DNA Kit (TIANGEN, China)를 이용해 전체 샘플을 대상으로 DNA를 추출 하였다. DNA 메타바코딩을 위해 COI 영역의 universal primer (mlCOIintF와 jgHCO2198; 증폭 길이 약 313 bp; Leray et al. 2013; Geller et al. 2013)에 MiSeq (Illumina, USA) 플랫폼의 어뎁터를 붙여 1차 PCR을 실시하였다. Universal primer의 Sequence (5’-3’)는 각각 mlCOIintF, GGWACWGGWTGAACWGTWTAYCCYCC 및 jgHCO2198, TAIACYTCIGGRTGICCRAARAAYCA였다.
COI 영역을 증폭하기 위해 rTaq Plus 5x PCR Master Mix (Elpisbio, Korea) 8 μl, universal primer 각 1 μl, 증류수 28 μl, 추출한 gDNA 2 μl를 첨가하여 총 반응 부피를 40 μl로 맞춘 후 중합효소연쇄반응(PCR)을 실시하였다. PCR 조건은 95°C에서 7분 denaturation 후, 95°C에서 30초 denaturation, 46°C에서 30초 annealing, 72°C에서 1분 extension 과정을 35회 반복 후, 72°C에서 5분간 final extension을 진행하였다. COI 영역의 증폭 여부를 확인하기 위해서 PCR 반응산물 2 μl를 2% TAE 아가로스 겔 상에서 전기영동을 하였다. 확인된 PCR 반응산물은 QIAquick PCR 정제 키트(QIAGEN, Germany)를 이용해 정제하였다.
1차 PCR 산물의 시료 구분을 위한 인덱스 부착을 위해 2차 PCR을 실시하였다(Illumina 2013). PCR 조건은 95°C에서 3분간 denaturation을 거친 후, 95°C에서 30초 denaturation, 46°C에서 30초 annealing, 72°C에서 30초 extension 과정을 8회 반복 후, 72°C에서 5분 final extension을 진행하였다.
2차 PCR 산물의 염기서열을 MiSeq (Illumina, USA)을 이용하여 대량의 염기서열을 추출하였다. 대량의 양방향 염기서열을 mothur (Schloss et al. 2009)의 MiSeq SOP (Kozich et al. 2013)에 따라 contigs를 만든 후 phylotype 방법으로 조작상 분류단위(operational taxonomic units, OTUs)를 만들었다. 이때 사용한 reference library와 taxonomy 파일은 NCBI/GenBank (Clark et al. 2016)에서 추출한 갑각류(Crustacean)의 COI 염기서열에서 동일한 것을 제거한 후 MAFFT (Katoh and Standley 2013)로 정렬하여 만들었다. Mothur (Schloss et al. 2009)를 이용하여 작성한 OTUs를 종으로 결정하기 위해 BLAST 검색(Altschul et al. 1990)으로 NCBI/GenBank (Clark et al. 2016)에서 유사 분류군의 염기서열을 추출한 후 OTU와 염기서열의 유사성이 99% 이상인 것을 종으로 결정하였다.
자료 분석
형태 분석과 DNA 메타바코딩 결과는 분류군 수준에 차이가 있어 확인된 종들을 강(Class) 또는 목(Order) 수준의 상위 분류군으로 묶고, 전체 출현 종 수에서 각 분류군에 속하는 종 수의 비율을 %로 표현하였다. 형태 분석에서 갑각류 외에도 다모류(Polychaeta), 이매패류(Bivalvia), 복족류(Gastropoda), 모악류(Chaetognatha), 미충류(Appendicularia)가 동정 되었고, DNA 메타바코딩에서는 이매패류와 거미불가사리류(Ophiuroidea)가 동정 되었지만, DNA 메타바코딩 분석 과정에서 갑각류만을 대상으로 하였기 때문에 두 방법의 비교를 위해 다른 분류군을 제외하였다. 형태 분석에서 요각류 유생(copepodite)과 난바다곤쟁이류 유생 단계(egg, nauplius, calyptopis, furcilia, juvenile)와 같은 생활사 단계는 비교 목적에 따라 제외하였다.
3. 결 과
형태 분석에 의해 총 17종의 갑각류 중형동물플랑크톤이 동정 되었으며, 하목(Infraorder) 수준의 1종, 과(Family) 수준의 3종, 속(Genus) 수준의 1종, 종(Species) 수준의 12종이었다(Table 1). DNA 메타바코딩(갑각류 COI reference library 사용)에 의해서는 총 18종이 동정 되었으며, 속 수준의 3종과 종 수준의 15종이었다. 분류군별로는 형태 분석의 경우 요각류(Copepoda) 11종, 단각류(Amphipoda) 2종, 십각류(Decapoda) 2종, 난바다곤쟁이류(Euphausiacea) 1종, 곤쟁이류(Mysida) 1종 이었다. DNA 메타바코딩은 지각류(Cladocera) 2종, 요각류 9종, 십각류 4종, 난바다곤쟁이 3종 이었다. 형태 분석에서는 지각류가 동정되지 않았고, DNA 메타바코딩으로는 단각류와 곤쟁이류가 동정되지 않았다.
Table 1.
Crustacean mesozooplankton taxa identified by morphological analysis and DNA metabarcoding
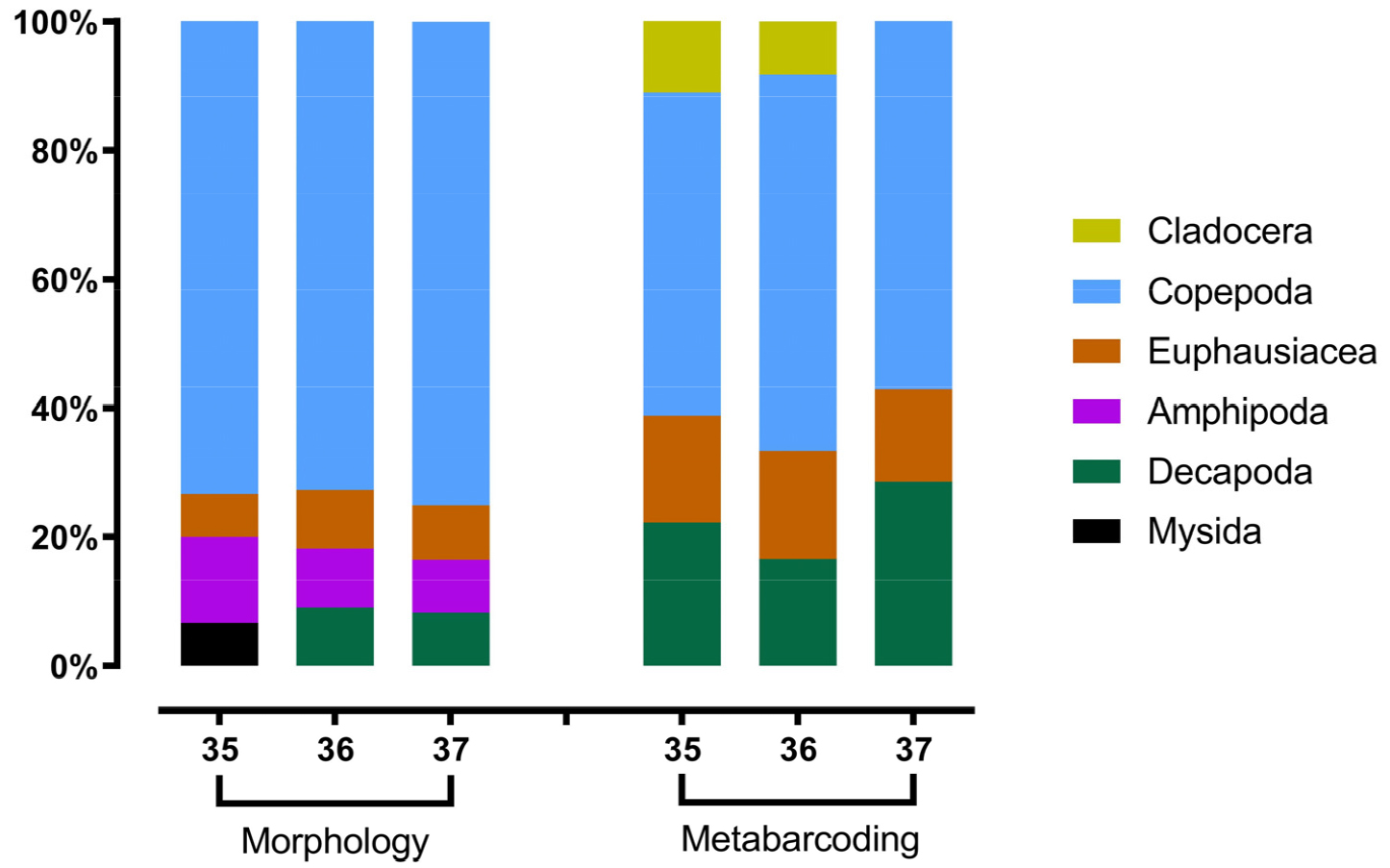
북위 35, 36, 37도 라인별 출현 분류군 수를 근거로 각 분류군의 자세한 구성을 보면(Fig. 2), 두 분석 방법 모두 가장 비율이 높은 분류군은 요각류로서 형태 분석에서는 전체 종 수의 약 73–75%를 차지하였으며, DNA 메타바코딩에서는 약 50–58%를 차지하였다. 형태 분석에서 단각류, 난바다곤쟁이류, 십각류의 비율은 비슷하였고, 곤쟁이류는 35도 라인에서만 출현하였다. DNA 메타바코딩에서는 형태 분석에 비해 난바다곤쟁이류와 십각류 종 수의 비율이 컸고, 지각류는 37도 라인에서 출현하지 않았다.
4. 고 찰
형태 분석에서 나타나지 않은 지각류 2종, 십각류 1종, 난바다곤쟁이 2종, 요각류 Acartia hudsonica가 DNA 메타바코딩으로 동정 되었다(Table 1). 형태 분석은 전체 시료에서 부차시료(subsample)를 취하는 과정에서 소수 출현한 종들이 무시될 수 있지만, DNA 메타바코딩은 시료 전체를 분석하기 때문에 소수종도 동정이 가능하다. DNA 메타바코딩은 해역에 유생으로만 소수 출현하여 형태 분석 결과 누락되었던 종을 찾아낼 수 있고(Lindeque et al. 2013), 현미경으로 관찰하기 어려운 소형동물플랑크톤이나 기생충도 동정이 가능하여서(Lindeque et al. 2013; Abad et al. 2016), 형태 분석으로 알 수 없었던 조사 해역의 숨겨진 다양성(hidden diversity)을 찾아낼 수 있다.
형태 분석이 십각류 유생을 목 수준의 상위 분류군까지 동정한데 비해, DNA 메타바코딩은 종 수준까지 동정이 가능하였다(Table 1). 상업적으로 이용되는 어류, 갑각류, 이매패류 등은 모두 초기 생활사 단계에서 플랑크톤 생활을 하는데, 이와 같은 일시플랑크톤은 유생기에 종별로 특징이 적어서 형태적인 동정이 어렵다. DNA 메타바코딩은 일시플랑크톤 동정에 효과적이어서, 이전 연구에서도 DNA 메타바코딩이 이매패류, 다모류, 복족류에 대해서 종 또는 속 수준까지 동정 가능하였고(Lindeque et al. 2013), 형태적으로 구분되지 않는 이매패류 유생에 대해 11개의 과를 찾아내기도 하였다(Hirai et al. 2017a). DNA 메타바코딩은 광범위한 해역에서 일시플랑크톤의 분포와 출현량 변동을 추적할 수 있기 때문에, 유영 또는 저서생활을 하는 상업종의 부유 유생기에 대한 연구에 활용될 수 있다.
DNA 메타바코딩은 성체가 아닌 알이나 유생이어도 종 동정이 가능하다는 강점이 있다. 그러나 이는 곧 이 자체로는 생활사 단계를 구분할 수 없다는 의미이기도 하다. 일례로 본 연구에서 난바다곤쟁이의 여러 생활사 단계(egg, nauplius, calyptopis, furcilia, juvenile)를 형태 분석으로 확인하였으나, DNA 메타바코딩으로 이를 구분하기 위해서는 별도의 표본 추출 과정이 필요하다. 동물플랑크톤의 생활사 구분은 생태 연구에서 중요한 부분으로, 개체군 구조와 세대 변동, 산란수와 생산력 등 많은 기능적 연구가 생활사 구분을 기초로 한다. 또한 동물플랑크톤의 분포와 출현량도 환경에 따라 성체와 유생이 다르게 나타날 수 있어서(Helle 2000; Yoon et al. 2000), 형태 분석이 종의 분포와 출현을 설명할 수 있는 여지를 준다. 예로서, 몬트레이 베이에서 요각류 Calanus pacificus가 연안보다 외해에서 많이 출현하는 것이 DNA 메타바코딩 결과로 나타났으나, 형태 분석으로 이들이 대부분 후기 유생(copepodite)인 것으로 확인되어(Harvey et al. 2017), 이 종의 생활사 기작(Peterson 1998)으로 이해할 수 있었다.
형태 분석에서 나타난 단각류와 곤쟁이류, 요각류 4종이 DNA 메타바코딩으로 확인되지 않았다(Table 1). 해양 동물플랑크톤은 다른 대형 동물들에 비해 유전자 데이터베이스가 부족한 편이어서, 해역의 데이터베이스 수준에 따라 동정의 정확성이 달라질 수 있다. 이전 연구에서 영국해협에 흔히 출현하던 요각류 Acartia clausi와 Oncaea 종이 데이터베이스 부족으로 동정되지 않거나(Lindeque et al. 2013), 요각류 Microsetella (Djurhuus et al. 2018)와, Clytemnestra (Schroeder et al. 2020)에 대한 데이터 부재도 있었다. 본 연구에서 DNA 메타바코딩으로 확인된 Acartia hongi (Table 1)는 처음에는 Acartia bifilosa로 동정 되었으나, 추가 분석결과 A.bifilosa COI 정보는 대서양 표본과 뚜렷하게 구분되었다. 따라서 처음의 A.bifilosa는 A. hongi의 COI 데이터베이스에 대한 종 동정 오류로 판단하였고, A. hongi의 COI 데이터베이스를 재등록 하기 위한 작업을 진행 중이다. 또한, 본 연구에서 DNA 메타바코딩으로만 동정된 요각류 Paraeuchaeta elongata는 북서태평양, 베링해, 오호츠크해, 동해 등지에서 출현하며(NIBR 2013), 강원도 이남의 해역에서는 출현이 보고된 바가 없어(Jeong et al. 2011), 같은 속의 다른 종일 가능성이 있으므로 추가적인 분석이 필요하다.
DNA 메타바코딩에서 DNA 추출, 증폭, pooling, 생물정보(bioinformatics) 과정에서도 편차가 생길 수 있다. 몬트레이 베이에서 28S rRNA 분석은 COI 분석보다 Calanus속의 요각류를 더 많이 동정하였으나 난바다곤쟁이류와 다모류에 대해서는 반대의 결과가 나타났고, 프라이머 선택 때문에 모악류와 미충류는 동정되지 않았다(Harvey et al. 2017). 본 연구에서 형태 분석으로 동정된 요각류 Ctenocalanus vanus, Labidocera euchaeta, Parvocalanus crassirostris, Oithona atlantica가 DNA 메타바코딩에서 확인되지 않은 이유도 PCR에 사용한 COI 프라이머와 이들의 COI 염기서열과의 결합력이 매우 낮기 때문으로 추정된다. 또한, DNA 증폭이 모든 분류군에서 동일하지 않으므로 동정에 영향을 줄 수 있고(Lindeque et al. 2013), 시퀀싱(sequencing) 종류의 선택이나 OTU (Operational Taxonomic Units) 결정 수준에 의해서도 분석 결과가 달라질 수 있다(Hirai et al. 2017b).
본 연구에서는 대상이 갑각류로 제한되었음에도 불구하고 형태 분석과 DNA 메타바코딩의 장단점이 잘 드러났고, 사후 검증을 통해 오류를 보완할 수 있는 기회가 되었다. 형태 분석은 생활사를 구분하고 정량화 할 수 있으므로, 해역의 동물플랑크톤 기능을 연구하는데 유용하다. DNA 메타바코딩은 보다 객관적으로 많은 분석을 빠르게 수행할 수 있으므로, 동물플랑크톤 다양성 파악에 강력한 도구가 될 수 있다. DNA 메타바코딩의 효과를 높이기 위해서는 데이터베이스 확충, 연구 해역의 분류군 탐지에 적합한 프라이머 개발, 분석 대상 표본의 제한(예, 알이나 유생 등)이 선행되어야 할 것으로 보인다. 향후 동물플랑크톤 연구에서 형태 분석과 메타바코딩을 활용한 DNA 분석을 병행할 경우 한가지 방법을 사용할 때보다 더 다양한 종 탐지와 생태적인 해석이 가능해지는 시너지 효과를 얻을 수 있을 것이다.